기 호 설 명
mRNA: messenger RNA
FACS: Fluorecence Activated Cell Sorter
RBD: Receptor Binding domain
CDR3: Complementarity-determining region
LNP: Lipid nanoparticle
1. 서 론
14세기부터 시작된 흑사병의 대유행을 기점으로 현재 SARS-CoV-2의 대유행에 이르기까지 병원성 박테리아 혹은 바이러스의 대유행이 지속적으로 발생하고 있다. 특히 2000년 이후에 유행된 감염병들은 사람과 동물이 같이 감염될 수 있는 인수공통감염병의 특징을 지니고 있다[1]. 이에 따라 World Health Organization (WHO)는 2018년 우리가 알지 못하는 많은 병원균 및 바이러스를 미지의 질병(Disease X)로 명명하고, 신·변종 감염병에 대한 연구 개발이 필요하다고 발표하였다[2].
이러한 바이러스는 DNA 합성, 유전자 편집 등의 분자생물학 및 합성생물학의 발전으로 인해 신·변종 바이러스 제조 및 재창조가 용이하게 되었고, 이를 악용하여 생물 테러의 심각성을 증가시킬 수 있는 잠재력이 있다[3]. 일례로, 바이러스 염기 서열 정보만으로 지구상에서 이미 멸종된 마두(horsepox) 바이러스를 다시 생산해 낸 연구가 보고되었다[4]. 따라서, 이처럼 생화학적 무기로 악용될 수 있는 신·변종 바이러스에 의한 감염병 예방 및 치료를 위해 백신과 치료제의 개발 필요성이 대두되고 있는 실정이다.
그러나 현재 SARS-CoV-2 바이러스의 대유행 상황에서도 볼 수 있듯, 긴급 사용 승인에도 불구하고 바이러스에 대한 백신 및 치료제의 품목 허가까지 최소 1년 이상의 기간이 소요되고 있다[5-7]. 이미 2020년 3월부터 급격히 확산되기 시작한 SARS-CoV-2 바이러스의 대유행은 2021년 절정을 이루며 수많은 변이를 양산하였으며, SARS-CoV-2의 염기서열 공개 및 백신 및 항체 치료제의 긴급 사용 승인에도 불구하고 대유행을 저지하지 못하고 있다[8-11]. 따라서 미래의 신·변종 대유행 바이러스 질병 발생 시 현재보다 획기적으로 신속한 백신 및 해독제 개발이 필수적이다.
현재 항체 치료제의 생산은 동물 세포 배양 및 정제 방법이 “gold standard” 방법으로서 수행되고 있다[12]. 이 방법들은 인체 내에서 안정된 항체를 생산할 수 있다는 점에서 강점이 있으나, 항체 치료제의 생산 과정에서 세포 배양액 자체에 바이러스나 다른 오염원의 오염 방지가 중요하고, 이를 위해 상당한 정도의 정제 과정이 필요하다. 또한 항체의 체외 생산 과정에서 post translational modification(PTM) 이 제대로 형성되어야 하는데, 각각의 과정에 해당하는 CMC (Chemistry, Manufacturing, and Control) 공정 요건이 까다롭기 때문에 질병에 대한 신속한 항체 제작에 어려움이 있다[13].
한편, 이러한 어려움을 극복하기 위해 mRNA에 기반한 항체 치료제가 개발되고 있다[14-16]. mRNA-항체 플랫폼은 LNP 제형화를 통해 체내에 쉽게 전달이 가능하며, 체내에서 전달 후 직접 항체가 생성되므로 세포 배양 시 발생할 수 있는 오염원을 최소화할 수 있다. 또한 체내에서 정확하게 PTM이 발생하므로 CMC 요건이 비교적 용이하다[17]. 이러한 장점으로 인해, mRNA-항체 후보 물질이 선별되면 이후 임상 시험에 신속하게 진입이 가능하다.
따라서 본 연구에서는 mRNA-항원 및 mRNA-항체 플랫폼, 그리고 항원 특이적 단일 기억 B 세포 분리를 이용하여 60일 이내에 비임상시험의 착수가 가능한 수준의 항체 치료제를 개발할 수 있는 항체 해독 플랫폼을 개발하였다. 본 연구에서는 SARS-CoV-2-delta-spike를 항원으로 이용하여, 항체 생성을 위해 기존 방식의 adjuvant-단백질 복합체가 아닌, 항원을 mRNA 백신의 형태로 제작하여 동물에 접종함으로서 적은 양으로도 높은 면역원성을 유도할 수 있도록 하여 4주 이내에 충분히 체내 항체 생성이 가능하도록 하였다. 접종된 mRNA 백신을 이용하여 동물의 Peripheral blood mononuclear cell(PBMC)와 splenocyte로부터 항원 특이적 B 세포를 분리하여 직접 항체를 분리함으로써 항체 확보 기간을 감소시켰다. 이렇게 확보된 항체 서열을 mRNA-항체 플랫폼에 적용함으로써 mRNA-항체의 생산 및 LNP 제형화를 통한 생산 공정 단계의 단순화를 통해 생산 속도를 신속화 하였다. 본 연구에서 기술된 항체 해독 플랫폼을 활용한다면, 40일 이내에 신속하게 신·변종 바이러스에 대한 백신 및 항체 치료제를 개발하고 생산할 수 있을 것이라 기대되는 바이다.
2. 실험재료 및 방법
2.1 실험동물
모든 동물실험은 대웅제약 동물실험윤리위원회(DWP-IACUC-23-041)의 승인을 받아 수행되었으며, 실험에 사용된 mouse는 오리엔트바이오(Seongnam-si, Gyeonggi-do, Korea)에서 구매하였다. 동물은 온도 22 ± 3 ℃, 상대습도 55 ± 15 %, 조명시간 12 시간, 조도 150 ∼ 300 lx 및 환기 횟수 10 ∼ 20 회/시간으로 설정하여 사육하였다. 사료는 mouse 용 고형사료(바이오피아 Co., Gunpo-si, Gyeonggi-do, Korea)를 전 실험기간 동안 자유 급여 하였으며, 물은 상수도수를 매일 자유 급여 하며 1일 1회 교체하였다.
2.2 SARS-CoV-2-delta mRNA 항원 발현 및 단일 기억 B 세포 분리를 위한 plasmid 제작
Mouse 내 mRNA 항원 접종 및 단일 기억 B 세포 분리를 위해, SARS-CoV-2-delta-Spike(GISAIS ID: EPI_ ISL_2100646) 및 RBD-Foldon-Fc 서열을 GISAID(https://www.gisaid.org)에서 확보하고, GenSmart 프로그램(https://www.genscript.com/gensmart-free-gene-codon-optim ization.html)을 이용하여 codon 최적화를 수행하여 유전자를 합성하였다(GenScript, NJ, USA). 합성된 SARS-CoV-2-delta-Spike 서열 및 RBD-Foldon 서열을 본 연구에서 자체 제작된 dual expression vector인 pDW202 plasmid에 삽입하기 위해, SARS-CoV-2-delta-Spike의 경우 Forward primer로서 5′-AACCCACCGGTGCCACCA TGTTTGTGTTCCT-3′ 서열을 가지는 oligonucleotide와 Reverse primer로서 5′-TGAGTGTCGACTCATCAGGGT GTAGTGCAGCTTTAC-3′ 서열을 가지는 oligonucleotide 를 제작하였고, RBD-Foldon의 경우 Forward primer로 5′-AACCCACCGGTGCCACCATGGACTGGACCTG-3′의 oligonucleotide와 Reverse primer로 5′-TGAGTGTCGAC TCACTTGCCAGGGGACAGTG-3′ oligomer를 사용하여 각각의 증폭 산물을 획득하였다. 제작된 amplicon들과 pDW202 vector는 Xba I과 BamH I 제한효소를 처리하여 37 ℃에서 1시간 반응시켰다. 증폭 산물의 경우 Qiagen(Venlo, Netherlands)의 PCR purification kit를 사용하여 제조업체의 방법에 따라 정제하였다. 제한효소 처리된 pDW202 vector는 1 % 한천 배지를 이용하여 젤 전기 영동을 통해 분리 및 정제하였다. 정제된 각각의 amplicon 및 pDW202 vector를 연결하기 위해, 1 μL 의 Quick ligase(New England Biolabs LTD., Ipswich, MA)와 1 X reaction buffer, amplicon 100 ng, pDW202 vector 100 ng이 포함된 혼합액 20 μL을 25 ℃에서 5분 동안 반응시킨 후, Escherichia coli DH5α(E. coli DH5α)에 형질 전환하였다. 형질 전환된 E. coli DH5α를 kanamycin Luria-Berani(LB) 한천 배지에 37 ℃에서 16시간 배양 후, E. coli DH5α colony를 획득하였다. 획득한 colony를 kanamycin이 포함된 LB 액체 배지에 접종하여 37 ℃에서 16 시간 배양한 후, 원심 분리하여 형질 전환된 E. coli DH5α 균을 획득하였다. 획득된 E. coli DH5α는 Qiagen의 DNA mini preparation kit 를 사용하여 제조업체의 방법에 따라 SARS-CoV-2- delta-Spike-pDW202 plasmid 및 RBD-Foldon-Fc-pDW202 plasmid를 획득하였다.
2.3 SARS-CoV-2-delta mRNA 항원 발현 및 단일 기억 B 세포 분리를 위한 plasmid 제작
단일 기억 B 세포 분리를 위해, RBD-Foldon 단백질을 발현하였다. RBD-Foldon-Fc-pDW202 plasmid를 HEK293FT 세포에 Lipofectamine 2000(ThermoFisher Scientific, Waltham, MA, USA)과 OptiMEM(ThermoFisher Scientific)을 사용하여 제조업체의 방법에 따라 형질 주입하여 발현시켰다.
RBD-Foldon 재조합 단백질의 정제는 Protein G bead (GE healthcare, Cicago, IL, USA)를 사용하여 제조사의 방법에 따라 수행하였다.
2.4 형광 표지 항원의 제조 및 FACS를 이용한 단일 기억 B 세포 분리
항원 특이적 B 세포 염색을 위해, 형광 표지 항원을 제조하였다. 정제된 재조합 항원인 RBD-Foldon 단백질에 EZ-Link Sulfo-NHS-LC-Biotylation kit(ThermoFisher Scientific)를 사용하여 제조사의 설명에 따라 biotinylation 을 수행하였다. 이렇게 biotin 표지된 항원을 streptavidin-APC(ThermoFisher Scientific)와 반응시켜 항원 특이적 B 세포 분리를 위한 형광 접합 항원 단백질을 제조하였다.
항원 특이적 단일 기억 B 세포는 면역화된 mouse의 비장세포로부터 얻었다. mouse로부터 적출한 비장세포를 PBS로 2회 세척 후, cell strainer에 통과시켜 불순물을 제거하였다. 이후, RBC lysis buffer(ThermoFisher Scientific)를 이용하여 적혈구를 제거하고 다시 PBS로 2회 세척 후 세포 수를 측정하여 세포의 수를 1 × 106 개로 조정하고 형광 염색을 수행하였다. 형광 표지된 시료에 anti-mouse CD16/32(BD bioscience, Franklin Lakes, NJ, USA), V450-anti-CD3(BD bioscience), V450-anti-CD4 (BD bioscience), V450-anti-CD8(BD bioscience), V450- anti-CD14(BD bioscience), PE-anti-CD19(BD bioscience), Live/dead staining dye(ThermoFisher Scientific) 및 형광 표지된 APC-RBD-Foldon을 첨가하여 20분간 얼음에서 염색하였다. 염색이 완료된 세포 중 Live, CD3-, CD4-, CD8-, CD14-, CD19+, RBD+ 항원 특이적 기억 B 세포를 FACSMelody Cell Sorter(BD bioscience) 장비를 이용하여 분리하였다. B 세포는 0.5 X PBS와 0.1 U RNase inhibitor로 구성된 buffer가 포함된 96 well plate 에 단일 분리 되었으며, reverse transcription 반응을 위해 - 80 ℃에 보관하였다.
2.5 cDNA 합성 및 IgG 유전자 증폭을 통한 신규 항체 후보군 variable region 확보 및 서열 확인
항원 특이적 단일 기억 B 세포의 cDNA 합성은 SuperScript III First Strand Synthesis kit(ThermoFisher Scientific)을 이용하여 제조업체의 설명에 따라 수행되었다. Ig 유전자는 이전에 보고된 방법을 이용하여 합성된 cDNA를 주형으로 하여 증폭하였다[18]. 각각의 Ig 유전자 증폭 산물은 1.5 % 한천 배지에서 젤 전기영동을 통해 확인 및 정제하였다. 정제된 각 Ig 유전자 증폭 산물은 Enzynomics 사의 TOPcloner™ TA core kit를 사용하여 제조업체의 방법에 따라 pTOP TA V2 vector에 cloning 하였다. Cloning된 각 Ig 유전자 증폭 산물은 sequencing 분석을 통해 서열을 확보하였다(Cosmogenetech, Seoul, Korea). 확보된 각 서열들은 IgBLAST(https://www.ncbi.nlm.nih.gov/igblast) 내 IMGT database를 활용하여 분석하고, 서열 상동성이 가장 높은 생식계열 V, D, J 유전자 구성을 확인하였다.
2.6 신규 항체 후보군 mRNA 합성을 위한 plasmid 구축
항원 특이적 단일 기억 B 세포 분리를 통해 획득한 신규 항체 후보군을 mRNA로 합성하기 위한 in vitro transcription plasmid를 제작하였다. 먼저, pFUSEss vector에 포함되어 있는 Heavy chain과 light chain의 각 constant region 절편을 Table 1의 primer를 이용하여 증폭한 후, 젤 전기영동을 통해 확인 및 정제하였다. 정제된 각 절편은 Xho I 제한효소 처리 후 실험 방법 section 2.2의 방법과 동일하게 pDW202 vector에 cloning 하여 Heavy chain과 Light chain 각각의 constant region이 포함된 backbone vector를 제작하였다(HC-pDW202, LC-pDW202).
Table 1.
Primer to obtain backbone plasmid
| Primer name | Sequences |
|---|---|
| Backbone-HC a-Forward primer | 5’-TTGCACTTGTCACGAATTCGA TATCTCGAGGCTGCTAGCACCA AGGGCCCAT-3’ |
| Bacbone-HC-Reverse primer | 5’-GAAGCATGGCCACCGAGGCT CCAGCCTCGATCATTTACCCGG AGACAGGGA-3’ |
| Backbone-LC b-Forward primer | 5’-TTGCACTTGTCACGAATTCGA TATCTCGAGGCATCAAACGTAC GGTGGCTGC-3’ |
| Backbone-LC-Reverse primer | 5’-GAAGCATGGCCACCGAGGCT CCAGCCTCGACTAACACTCTCC CCTGTTGA-3’ |
각각의 신규 항체 후보군 절편들에 대해 DW-S-Ab-01, DW-S-Ab-02, DW-S-Ab-09, DW-S-Ab-10, DW-S-Ab-11, DW-S-Ab-21, DW-S-Ab-25, DW-S-Ab-31로 각각 명명하였고, Table 2의 primer를 이용하여 PCR 반응을 통해 증폭 산물을 획득하였다. 즉, Heavy chain의 경우 DW-S-Ab-01은 primer 1과 primer 2를, DW-S-Ab-02, 21, 25는 primer 1과 3을, DW-S-Ab-09는 primer 1과 5를, DW-S-Ab-10, 31은 primer 1과 6을, DW-S-Ab-11의 경우에는 primer 1과 7을 각각 이용하여 증폭 산물을 획득하였다. Light chain의 경우 DW-S-Ab-01은 primer 8과 9를, DW-S-Ab-02는 primer 10과 11을, DW-S-Ab-09는 primer 12와 13을, DW-S-Ab-10은 primer 14와 9를, DW-S-Ab-11은 primer 8과 11을, DW-S-Ab-21은 primer 15와 9를, DW-S-Ab-25는 primer 16과 17을, DW-S-Ab-31은 primer 8과 17 set를 각각 활용하여 증폭 산물을 획득하였다. 획득한 각각의 신규 항체 후보군 절편들에 대해서, Heavy chain의 variable region 절편의 경우 EcoR I 및 Nhe I 제한효소를 처리하고, light chain의 variable region 절편의 경우 EcoR I 및 Bswi I 제한효소를 처리하여 backbone vector에 cloning하여 최종적으로 heavy chain 및 light chain의 in vitro transcription vector를 획득하였다.
Table 2.
Primer sequences for variable region of novel antibody candidates
2.7 항원 및 신규 항체 후보군 mRNA 합성
In vitro transcription 반응을 통해 mRNA를 획득하기 위해, SARS-CoV-2-delta-Spike-pDW202 plasmid를 Sap I 제한효소 처리하여 plasmid를 선형화하였다. 선형화된 plasmid는 Qiagen의 DNA Maxi preparation kit를 사용하여 제조업체의 방법에 따라 정제되었다. SARS-CoV-2-delta-Spike의 mRNA 합성을 위해, 선형화된 plasmid 20 μg, AG Cap(Trilink, San Diego, CA, USA) 8 mM, 각 rNTP 10 mM, 1X reaction buffer, MEGAscript T7 enzyme mix(ThermoFisher Scientific) 40 μL가 포함된 400 μL 혼합액을 37 ℃에서 4 시간 반응시켰다. 이후 DNase I(ThermoFisher Scientific) 40 μL를 처리하여 15 분 동안 37 ℃에서 반응한 후, LiCl 정제 방법을 이용하여 합성된 mRNA를 정제하였다[19].
2.8 LNP-mRNA 제형화
LNP-mRNA 제형화를 위해, 모든 제제는 먼저 지질(이온화 지질, Cholesterol, DSPC, PEG2000-DMG)을 에탄올에 몰 비율이 각각 50 %, 38.5 %, 10 %, 1.5 % 이 되도록 용해시켜 제제화하였다[20,21]. 신규 항체 후보군의 mRNA 경우 heavy chain과 light chain mRNA 의 몰 비율을 1:1로 혼합하였다. 그 후, 지질 혼합물을 pH 4.5인 0.1 M sodium citrate 완충액과 혼합된 mRNA 수용액에 아민/인산염 비율(N/P)이 6, 그리고 1:3 에탄올/물 비율이 되도록 microfluidic mixing cartridge(NanoAssemblr® Ignite Cartridges, Precision Nanosystems Inc., Vancouver, Canada)를 이용하여 혼합하였다[20]. 생성된 혼합물은 10,000 배 이상 부피의 pH 7.4 PBS를 이용하여 하루 동안 투석하였다. 이렇게 생성된 LNP-mRNA는 Malvern Zetasizer Nano ZS를 사용하여 동적 광 산란을 통해 입자 크기를 측정하였다.
2.9 LNP-mRNA 백신 접종
신규 항체 발굴을 위한 항체 형성 확인 및 투여 경로 결정을 위해, 정상 Balb/C 6주령 mouse에 LNP-mRNA 항원 백신 0.5 mpk의 양을 I.V., I.M. 및 I.P. 투여 경로를 통해 투여하였다. 2주 후 각 경로를 통해 0.5 mpk의 양을 추가 투여하고, 25일차에 다시 0.5 mpk 의 양을 추가 투여하였다. 주차 별로 경정맥 채혈을 통해 혈액을 채취하였으며, 4주 차는 전혈 채혈하였다. 혈청은 4 ℃, 10,000 rpm으로 30분간 원심 분리하여 획득하였다. 획득한 혈청 시료와 Human IgG ELISA Kit(ThermoFisher Scientific)을 사용하여 제조사의 방법에 따라 혈청 시료 내 항체의 농도를 측정하였다.
2.10 신규 항체 후보군의 약동학(Pharmacokinetics, PK) 측정
확보한 mRNA-신규 항체 후보군의 생체 내 효능을 확인하기 위해, 약동학 실험을 수행하였다. 6주령 C57BL/6 mouse(Orientbio)의 꼬리 정맥을 통해, 2 mpk 의 LNP-mRNA 신규 항체를 주사하고 0 h, 3 h, 6 h, 12 h, 24 h, 48 h가 경과한 시점에 경정맥 채혈하였다. 혈청은 혈액을 4 ℃, 10,000 rpm으로 30분간 원심 분리하여 획득하였다. 획득한 혈청 시료와 Human IgG ELISA Kit(ThermoFisher Scientific)을 사용하여 제조사의 방법에 따라 혈청 시료 내 항체의 농도를 측정하였다.
3. 결 과
3.1 신속 신규 항체 후보군을 획득하기 위한 항원 특이적 단일 기억 B 세포 분리 방법의 개념
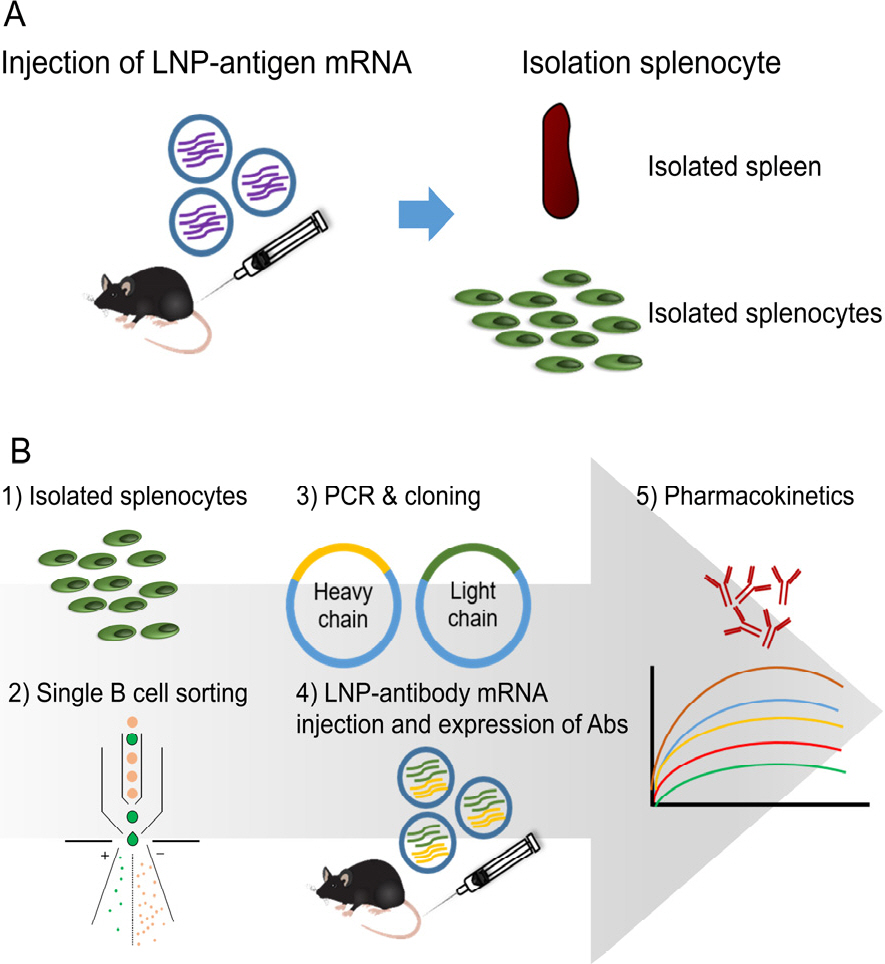
본 연구에서 개발된 항원 특이적 단일 기억 B 세포 분리 방법은 신종 바이러스에 대한 항체를 신속하게 획득하기 위한 방법이다. 인간의 immunoglobulin 유전자는 V(D)J recombination을 통해 항체를 만들어 내는데, 다른 세포와는 달리 적응 면역 시스템의 B 세포 발달 과정에서는 부모의 생식세포로부터 유전된 germline immunoglobulin 유전자가 재배열을 거치게 되며, 이러한 과정을 감수 분열 재조합과 구별하기 위해 somatic recombination이라 칭한다. B 세포의 초기 발달에서 somatic recombination을 통해 항체의 다양성이 만들어지며, 특정 항원이 우리 몸 안에 들어오게 되면 germinal center에서 somatic hypermutation이 일어나게 된다. Somatic hypermutation은 활성화된 T 세포의 신호가 존재할 때, 항원 활성적인 germinal center의 B 세포에서 발생하며, 항원에 대해 더욱 선택적인 항체를 생성하게 된다. 이러한 과정을 통해 부모에서 유전된 germline 항체 유전자로부터 항원 특이적인 새로운 항체가 만들어지게 되는 것이다.
이러한 B cell의 발달을 통한 항체 생성 방법을 이용, 단일 기억 B 세포 분리 방법을 통해 새로운 항체를 개발하는 시스템을 구축하였다(Fig. 1). 먼저 항원으로서 SARS-CoV-2-delta-Spike 단백질을 활용하였고, 항원이 체내에서 발현되도록 mRNA-항원을 mouse에 예방 접종하여 항원 특이적 항체를 생성하는 B 세포 발달을 유도하였다(Fig. 1A). 주입한 mRNA-항원에 의해 항원 특이적 항체 서열을 가지는 B 세포는 FACS cell sorter 장비를 통해 단일 세포로 분리되고 B 세포의 RNA 추출 및 cDNA 합성, PCR 반응을 통해 항원 특이적 항체의 variable region 서열 확보가 가능하다 (Fig. 1B).
3.2 mRNA-SARS-CoV-2-delta-spike(RBD-fd) 항원 접종을 통한 항체 생성 확인
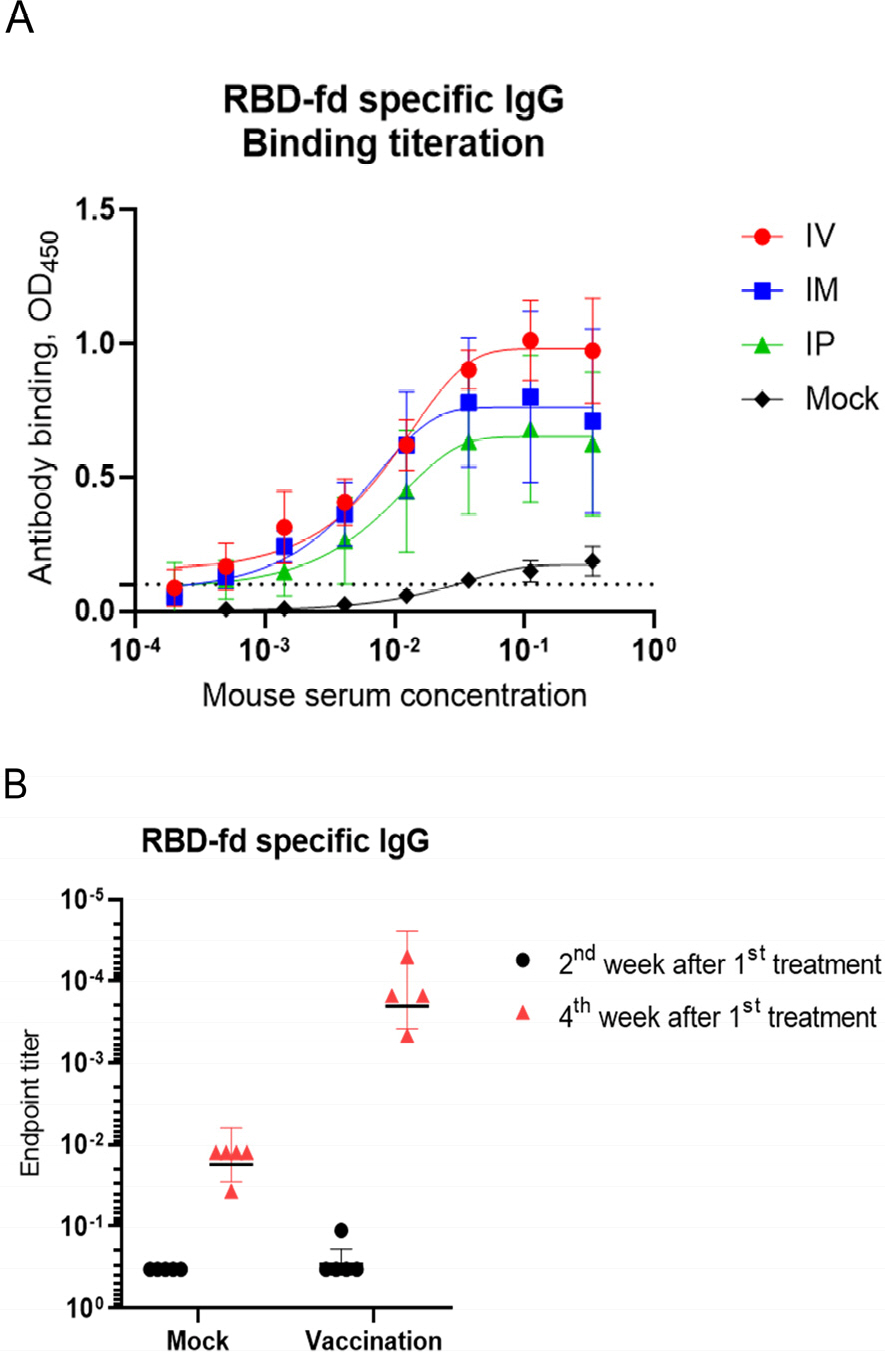
항원인 SARS-CoV-2-delta-spike의 mRNA를 합성하여 LNP-mRNA로 제형화하고, 이를 접종한 C57BL/6 mouse 에서 각 주차 별로 혈청을 획득하였다. 획득한 각 주차 별 혈청으로 ELISA를 통해 endpoint dilution titration assay를 진행하여 항체가 생성된 개체를 확인하였다(Fig. 2). 그 결과, 모든 투여 경로에서 항체가 생성되는 것이 확인되었고, 특히 I.V. 투여 경로가 I.P. 및 I.M. 경로에 비해 항체가 잘 생성되는 것을 확인하였다(Fig. 2A). 또한, 항체 생성 기간을 확인하기 위해 ELISA를 이용한 각 주차 별 항체 농도 확인 결과, 접종 후 4 주차에 항체가 가장 많이 생성되는 것을 확인하였다(Fig. 2B).
3.3 항원 특이적 단일 기억 B 세포 분리 방법을 통한 신규 항체 후보군 획득
신규 항체 후보군을 확보하기 위해, 항원인 LNP-SARS-CoV-2-delta-Spike-mRNA를 C57BL/6 mouse에 I.V. 접종 후 4주 차에 비장을 적출하였다. LIVE/DEAD 염색을 통해 약 5000 개의 B 세포를 1차적으로 분리하고, CD4-, CD8-, CD14- 특이적 B 세포를 분리한 후, CD19+/RBD-foldon-APC+에 특이적인 B 세포를 분리한 결과, 약 16개의 항원 특이적 B 세포를 획득하였다(Fig. 3A). 16개의 항원 특이적 B 세포에 대한 RNA를 추출한 후 Reverse transcription PCR 및 Nested PCR 수행 결과, 13 종의 항체가 확보되었음을 확인할 수 있었다(Fig. 3B). 13종의 항체에 대해 서열 분석을 수행한 결과, 중간에 종결 코돈이 존재하지 않는 온전한 8 종의 신규 항체 후보군을 최종적으로 획득하였다(Table 3). 8종의 신규 항체 후보군에 대해 germline mutation 서열 분석 결과, antigen 결합 부위인 CDR3 region에서 높은 variation을 보이는 것을 확인하였다(Table 4). CDR3 region의 경우 항원에 직접적으로 결합력에 영향을 미치는 부분이기 때문에, 이와 같은 CDR3 region의 mutation이 항원의 특이성을 더욱 높일 것으로 추정되었다.
Fig. 3.
Screening results for antibody candidates using antigen-specific single memory B cell sorting method, A) Result for antigen specific single memory B cell separation, B) Fragments for heavy and light chain of novel antibody candidates

Table 3.
sequencing result for novel antibody candidates using IMGT
Table 4.
Comparison for CDR3 sequences of novel antibody candidates
| Antibody candidates | Germline sequences | Novel sequence | ||
|---|---|---|---|---|
| Heavy chain | Light chain | Heavy chain | Light chain | |
| DW-S-Ab-01 | ARYYGSS YYFDY | QHFWG TPWT | ARG a YYG SSYYFDY | QHFWG TPRT |
| DW-S-Ab-02 | TSTMVTW FAY | GQSYSY PLT | T b LYPGV AY | GQSYSY PPT |
| DW-S-Ab-09 | ARFITTVV AYWYFD V | QQWSSN PPYT | ARS a PYYY GSSGGYF DV | QQWSSN HMYT |
| DW-S-Ab-10 | ARLTGYF DY | QQYSGY PLW | ARG a GLR YFDY | QQYSGY PLT |
| DW-S-Ab-11 | ARSTMVT YYAMDY | QHHYGT PLT | ARAWFPY YHAMDY | QHHYGT PLT |
| DW-S-Ab-21 | ARLTGWF AY | QQGNTL PWT | ARKS a LTA WFAY | QQGNTL PWT |
| DW-S-Ab-25 | ARFITTVV AWFAY | LQYDEF PYT | AR b SDYY GSLFAY | LQYDEF PYT |
| DW-S-Ab-31 | ARFITTVV AYFDY | QQFTSS PSYT | ARSGS b SY FDY | QQFTSS PSbT |
3.4 신규 항체 후보군의 약동학 측정
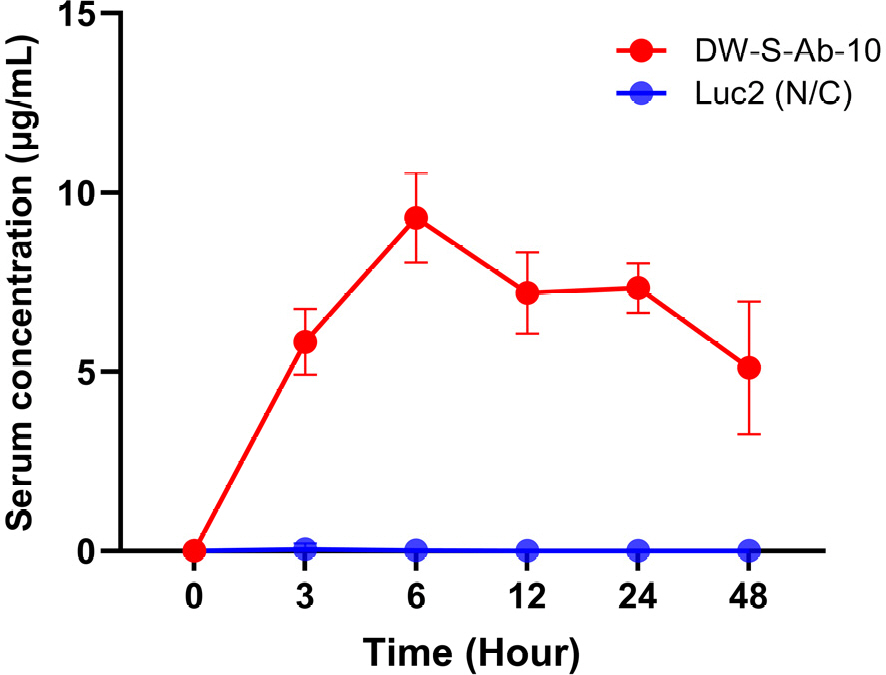
약동학 측정을 위해, 항원인 SARS-CoV-2-delta spike 단백질에 결합하지 않는 대조군으로서 luciferase 유전자(Luc2)와, 항원 특이적 단일 기억 B 세포 분리를 통해 획득한 신규 항체 후보군 8종 중 DW-S-Ab-10에 대해 mRNA를 합성하고 LNP로 제형화하였다. 제형화된 각 LNP-mRNA의 particle size 및 zeta potential 측정 결과, Table 5와 같이 약 86 nm, Polydispersity(PI)는 약 0.1로 LNP-mRNA가 제형화 되었음을 확인할 수 있었다. 제형화된 2 mg/kg의 LNP-mRNA를 C57BL/6 마우스에 꼬리 정맥으로 투여한 후, 0 시간부터 48 시간 동안 시간별로 채혈하여 신규 항체 후보군의 약동학을 측정하였다. 그 결과, 6 시간 내외로 10 μg/mL 정도의 항체 생성 수준에 도달하고, 48시간까지 약 5 μg/mL의 농도 수준의 항체가 생성되는 것을 확인하였다(Fig. 4). 이를 통해, 항원 특이적 단일 기억 B 세포 분리를 통해 획득한 신규 항체 후보군을 LNP-mRNA 로 제형화하여 백신 접종하였을 때, 체내에서 항체로 발현되어 항원과의 특이적 결합을 통해 SARS-CoV-2- delta 바이러스에 대한 중화 항체로서 활용도가 높을 것으로 예상되었다.
4. 결론 및 고찰
14 세기 흑사병 출현을 기점으로 지속적으로 발생하는 신·변종 감염병 유발 바이러스는 지구 온난화 등 환경 변화와 교통 수단의 발달 등으로 빠르게 확산되고 있는 추세이다. 심지어 분자생물학 및 합성생물학 분야의 발전으로 인해 바이러스의 염기서열을 쉽게 획득할 수 있으며, 획득한 바이러스의 염기서열을 기반으로 병원성 바이러스를 재창조하여 생화학 무기로서의 악용이 가능할 것으로 예상된다. 따라서, 바이러스 유래의 신·변종 감염병 발생의 신속한 대처 뿐 아니라 병원성 바이러스의 재창조라는 위협적인 요소를 미연에 방지하기 위해서 단기간에 백신 및 해독제를 개발해야 할 필요성이 대두되고 있다.
본 연구에서는 항원 특이적 단일 기억 B 세포 분리 방법을 통해 신속하게 항체를 획득할 수 있는 항체 해독 플랫폼을 개발하였다. Corona delta virus의 spike 단백질을 항원으로 이용하여 mRNA 플랫폼 기반 면역 접종을 수행함으로서, 항원 특이적 항체를 생성하는 B 세포 발달을 유도하였다. 주입한 mRNA-항원에 의해 마우스의 germline center B 세포에서는 somatic hypermutation을 통해 항원 특이적 항체 서열이 생성된다. 이후 항원 특이적 단일 기억 B 세포 분리 및 항체 유전자 데이터베이스를 활용하여, 발굴된 항체 유전자 서열을 분석함으로써 새로운 항체 후보군을 확보하였다. 확보된 항체 후보군 서열을 토대로 mRNA 를 in vitro transcription 반응을 통해 합성하고, 이를 LNP-mRNA로 제형화 하였다. 제형화된 항체 후보군의 LNP-mRNA 는 mouse 체내 접종 후 약동학 측정을 통해 체내 발현량 확인 및 항원인 corona delta 바이러스의 spike 단백질과의 결합을 확인함으로써 SARS-CoV-2-delta 바이러스에 대한 중화 항체로서 활용될 수 있는 가능성을 제시하였다. 본 연구에서 개발된 항체 해독 플랫폼을 통해, 백신 접종 후 약 40일 만에 신·변종 바이러스에 대한 항체 해독제 확보가 가능함을 확인할 수 있었으며, 이를 통해 항체 및 해독제를 신속하게 개발할 수 있을 것으로 기대된다.